Groupe Quinones Respiratoires
- Membres & Collaborateurs
- Le groupe Quinones Respiratoires
- Historique
- Projets
- Financements
- Emplois
- Publications
Membres du groupe
- Fabien Pierrel (Chercheur CNRS) co-responsable de Groupe - Fabien.Pierrel [at] univ-grenoble-alpes.fr
- Ludovic Pelosi (Enseignant-Chercheur UGA), co-responsable de Groupe - Ludovic.Pelosi [at] univ-grenoble-alpes.fr
- Amélie Amblard (Assistante Technique, UGA)
- Mahmoud Hajj Chehade (Enseignant-Chercheur CDD UGA)
- Katayoun Kazemzadeh (Doctorante CNRS ANR)
- Sophie-Carole Chobert (Doctorante CNRS ANR)
- Morgane Roger Margueritat (Doctorante UGA)
- Emilie Launay (Doctorante UGA)
- Laura Flandrin (Assistante Ingénieure, CDD CNRS)
Collaborateurs au sein de l’équipe TrEE
- Sophie Abby (Chercheuse CNRS) - Sophie.Abby [at] univ-grenoble-alpes.fr
- Ivan Junier (Chercheur CNRS) - Ivan.Junier [at] univ-grenoble-alpes.fr
- Nelle Varoquaux (Chercheuse CNRS) - Nelle.Varoquaux [at] univ-grenoble-alpes.fr
Collaborations
- Marc Fontecave (Collège de France, Paris, France)
- Frédéric Barras (Aix-Marseille Université, France)
- Catherine Clarke (Department of Chemistry and Biochemistry, UCLA, USA)
- Leonardo Salviati (University of Padova, Italy)
- Dave Pagliarini (University of Wisconsin, Madison, USA)
- Mickael Cohen (IBPC, Paris, France)
- Isabelle André (TBI, Toulouse, France)
- Volker Wendisch (Bielefeld University, Germany)
- Roland Lill (Marburg University, Germany)
- Yvain Nicolet (IBS, Grenoble, France)
Quinones Respiratoires ↗
La majorité de l’énergie des cellules bactériennes et eucaryotes provient de chaînes de transfert d’électrons qui permettent in fine la synthèse d’ATP par l’ATP synthase. Le fonctionnement de ces chaînes de transfert d’électrons (photosynthétiques ou respiratoires) dépend de petites molécules lipidiques, appelées quinones isoprénoïdes (Figure 1a). Ces quinones isoprénoïdes sont donc essentielles aux processus bioénergétiques et au métabolisme de quasiment tous les êtres vivants.
Les quinones isoprénoïdes les plus communes sont la plastoquinone (PQ, organismes photosynthétiques), la ménaquinone (MK, bactéries et archées) et l’ubiquinone (UQ, protéobactéries et eucaryotes), encore appelé coenzyme Q (Figure 1b).
✓ Comment les quinones sont-elles synthétisées dans les cellules ?
✓ Dans quels processus physiologiques interviennent les quinones ?
✓ Comment le métabolisme et les quinones ont évolué depuis les cellules primitives ?
Voici les questions fondamentales qui nous animent et auxquelles nous tentons de répondre en étudiant les microorganismes par une combinaison d’approches expérimentales (génétique, biochimie, biologie moléculaire) et bio-informatiques à grande échelle (génomique et phylogénétique).
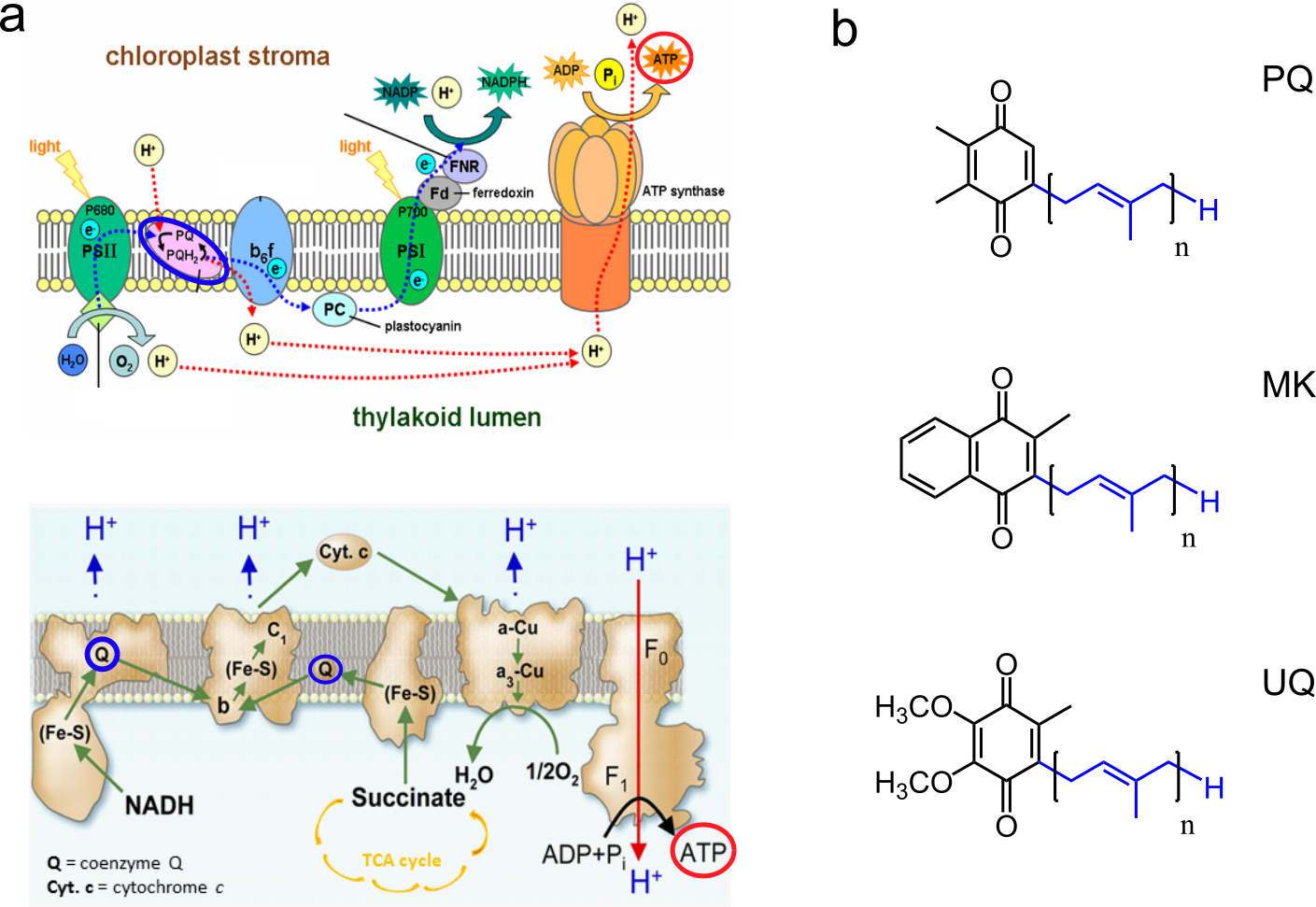
Figure 1 : a) Chaînes de transfert d’électrons photosynthétiques et respiratoires avec les quinones entourées en bleu et l’ATP en rouge.
b) Structure chimique des quinones les plus communes avec la chaîne polyprényle de longueur variable en bleu (n=6-12 en fonction des organismes).
Historique de nos contributions ↗
Au cours des 10 dernières années, nous avons largement contribué à une meilleure compréhension des voies de biosynthèse de l’ubiquinone (UQ) chez des organismes modèles tels que la levure Saccharomyces cerevisiae et la protéobactérie Escherichia coli. Nous avons notamment identifié et caractérisé de nouveaux gènes (Figure 2) et montré que des analogues synthétiques du précurseur de l’UQ (par exemple l’acide vanillique) peuvent complémenter des déficiences de certaines étapes de biosynthèse, ce qui a ouvert de nouvelles perspectives pour le traitement des déficiences primaire en UQ (coenzyme Q) chez l’homme.
Enfin, nous avons montré que l’UQ est importante pour le métabolisme d’organismes pathogènes tels que Salmonella typhimurium, Pseudomonas aeruginosa et Francisella tularensis, suggérant que la voie de biosynthèse de l’UQ pourrait être une cible antibactérienne.
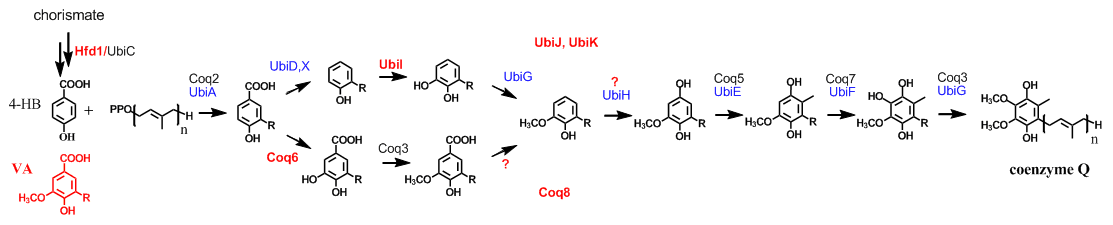
Figure 2 : Voies de biosynthèse du coenzyme Q chez E. coli (noms des protéines en bleu) et chez S. cerevisiae (en noir), n=6-8. Les contributions significatives de notre groupe sont en rouge. Coq8, UbiJ et UbiK participent à la biosynthèse de Q sans catalyser de réactions chimiques. 4-HB, acide 4-hydroxybenzoïque et VA, acide vanillique.
Certains projets que nous développons actuellement sont décrits ci-dessous.
I- Organisation supramoléculaire de la biosynthèse de UQ ↗
Projet soutenu par l’EUR-CBS UGA
La biosynthèse de l’UQ nécessite 7 modifications consécutives d’intermédiaires extrêmement hydrophobes. En 2019, nous avons montré que 7 protéines Ubi forment un métabolon stable d’environ 1 MDa qui catalyse les six dernières étapes de la biosynthèse de l’UQ chez E. coli (Figure 3). Outre les 5 enzymes responsables des réactions chimiques, le complexe multiprotéique contient les protéines UbiJ et UbiK qui lient les intermédiaires hydrophobes de biosynthèse.
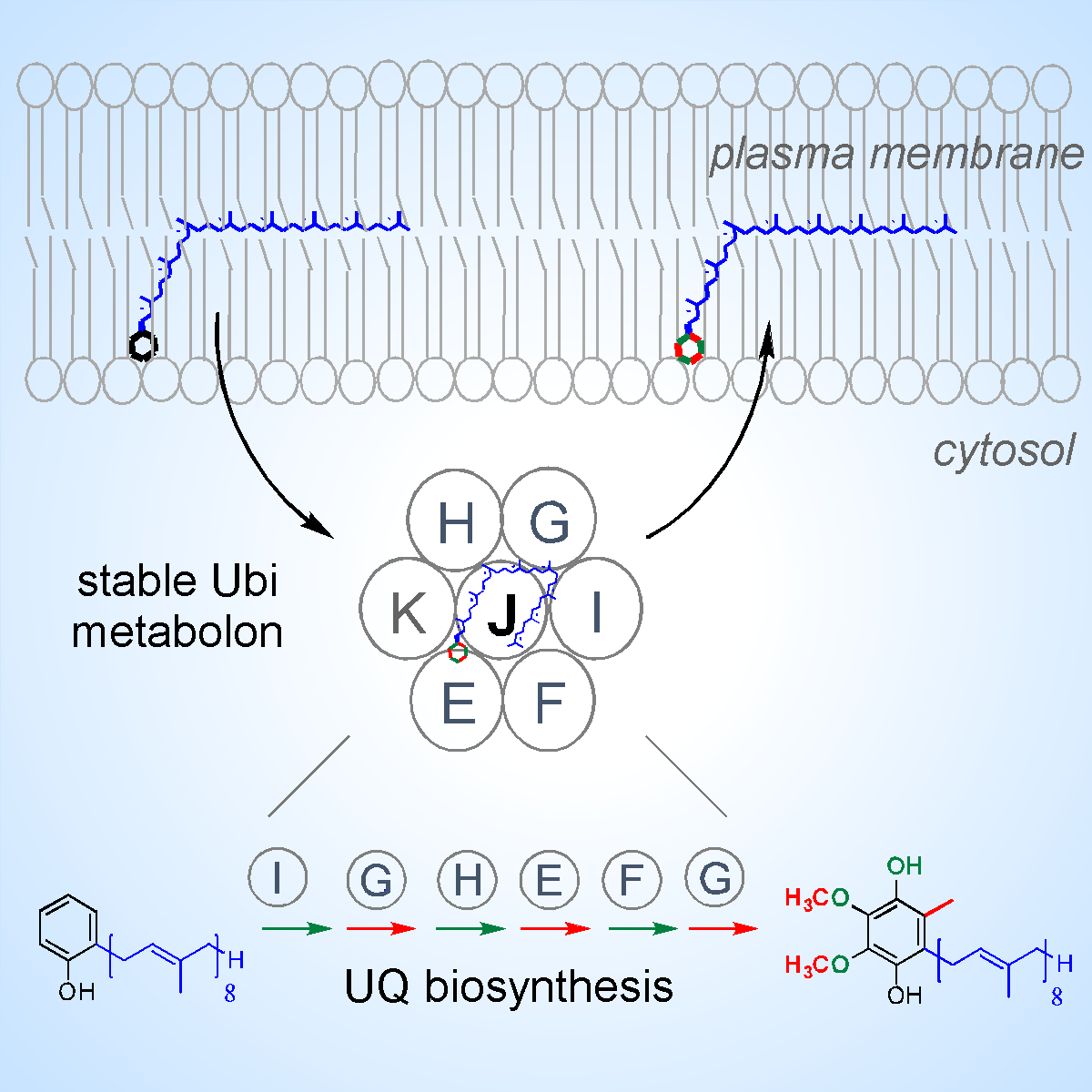
Figure 3 : Le métabolon Ubi, composé de sept protéines, catalyse les six dernières étapes de la voie de biosynthèse du coenzyme Q chez E. coli.
Nous travaillons à produire et à purifier le complexe Ubi afin de déterminer sa structure et ainsi comprendre comment la cellule orchestre la biosynthèse de UQ à l’échelle moléculaire. Pour cela, nous avons construit un système de surproduction du complexe Ubi grâce à une technologie de clonage modulaire et nous proposons de résoudre en collaboration avec l’IBS de Grenoble la structure du complexe purifié par cryomicroscopie électronique. Cette partie du projet a récemment était lauréat d’un appel d’offre « École Universitaire de recherche » en Chimie Biologie et Santé de l’Université Grenoble Alpes (CBH Graduate School) coordonné par L. Pelosi.
Le complexe Ubi existe-t-il chez d’autres bactéries ? Comment a-t-il évolué sachant que les 7 protéines du complexe Ubi d’E. coli ne sont pas toutes strictement conservées ? Nous abordons ces questions en collaboration avec des chercheurs et chercheuses de l’équipe TrEE (Ivan Junier et Sophie Abby) via des analyses génomiques et co-évolutives.
II- Découverte d’une voie de production de UQ indépendante d’O2 ↗
Projet soutenu par l’ANR
Ce projet est mené en collaboration avec les groupes de Frédéric Barras (Institut Pasteur, Paris) et Marc Fontecave (Collège de France, Paris) dans le cadre des projets ANR « (An)aeroUbi » (ANR-15-CE11-0001) et « O2-taboo » (ANR-19-CE44-0014) coordonné par F. Pierrel.
La voie de synthèse classique de l’UQ nécessite la présence de dioxygène (O2) qui est utilisé dans trois étapes d’hydroxylation (Figure 4). Or, de nombreuses bactéries, appelées anaérobies facultatives, sont capables de se développer en présence et en absence d’O2 et doivent ainsi adapter leur métabolisme. Pendant le projet ANR (An)aeroUbi (2015-2020), nous avons découvert une nouvelle voie de production de UQ indépendante d’O2. Nous avons identifié trois nouveaux gènes d’E. coli, ubiT, ubiU et ubiV, qui interviennent spécifiquement dans la voie indépendante d’O2 et qui participent probablement aux étapes d’hydroxylation, les seules qui différent de la voie dépendante d’O2 (Figure 4).
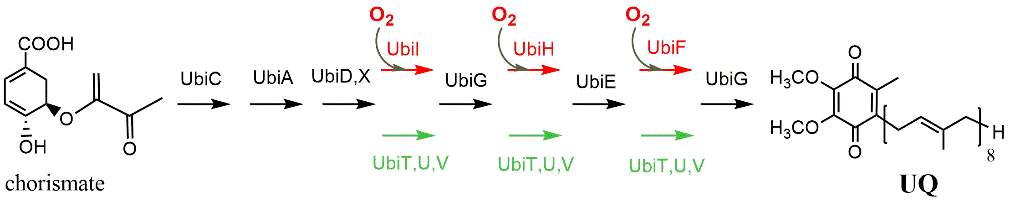
Figure 4 : Biosynthèse de l’ubiquinone (UQ) chez E. coli par la voie dépendante d’O2 (rouge) et par la voie indépendante d’O2 (vert) nouvellement identifiée. Les protéines en noir sont communes aux deux voies.
Nos analyses bio-informatiques ont établi que les gènes ubiT, -U, -V sont conservés chez de nombreuses protéobactéries, suggérant que ces espèces sont elles aussi capables de synthétiser UQ en anaérobiose, ce qui était jusqu’ici insoupçonné. Nous avons établi l’importance physiologique de cette nouvelle voie de synthèse de UQ chez Pseudomonas aeruginosa, en montrant que les gènes ubiT, -U, -V sont essentiels à la dénitrification, un métabolisme anaérobie largement utilisé par P. aeruginosa dans les poumons de patients atteints de mucoviscidose.
Dans le cadre du projet O2-taboo (2019-2023), nous cherchons à comprendre la régulation des voies O2-dépendante et O2-indépendante de biosynthèse de UQ, à définir le mécanisme des protéines UbiU et UbiV qui sont vraisemblablement les prototypes d’une nouvelle famille d’hydroxylases, et à déterminer l’importance physiologique de UQ en anaérobiose, notamment en étudiant la capacité de souches modifiées d’E. coli à coloniser l’intestin de souris. Ce projet fait l’objet de la thèse de Katayoun Kazemzadeh.
Enfin, notre découverte d’une voie de biosynthèse de l’UQ indépendante d’O2 questionne l’hypothèse actuelle de l’émergence de la voie UQ lors de l’oxygénation de l’atmosphère, il y a plus de 2 milliards d’années. Ainsi, grâce à des approches phylogénétiques, nous essayons d’établir laquelle des voies de biosynthèse de l’UQ (dépendante ou indépendante d’O2) est apparue en premier.
III- Évolution de la régio-sélectivité des hydroxylases de la voie de biosynthèse de UQ chez les protéobactéries ↗
Projet soutenu par l’ANR (ANR-19-CE45-0013, projet DEEPEN coordonné par Ivan Junier)
Ce projet est développé en collaboration avec Ivan Junier, Sophie Abby et Nelle Varoquaux de l’équipe TrEE, et avec le groupe d'Isabelle André (TBI, Toulouse).
Nous avons découvert deux nouvelles clades d’hydroxylases impliquées dans la biosynthèse de UQ (UbiM and UbiL, mSystems 2016) (Figure 5) et montré que les protéobactéries ont évolué différemment pour catalyser les trois réactions d’hydroxylation nécessaires à la biosynthèse de UQ: certaines bactéries possède une seule hydroxylase avec une régio-sélectivité large (qui peut hydroxyler plusieurs positions du noyau aromatique), alors que d’autres possèdent trois hydroxylases régio-sélectives (qui hydroxylent chacune une position spécifique). En combinant approches expérimentales et bioinfomatiques (phylogénie, modélisation et analyses de coévolution), nous identifions et étudions les UQ hydroxylases présentent dans l'ensemble des génomes bactériens afin de comprendre leur évolution et de découvrir les éléments contrôlant la régiosélectivité.
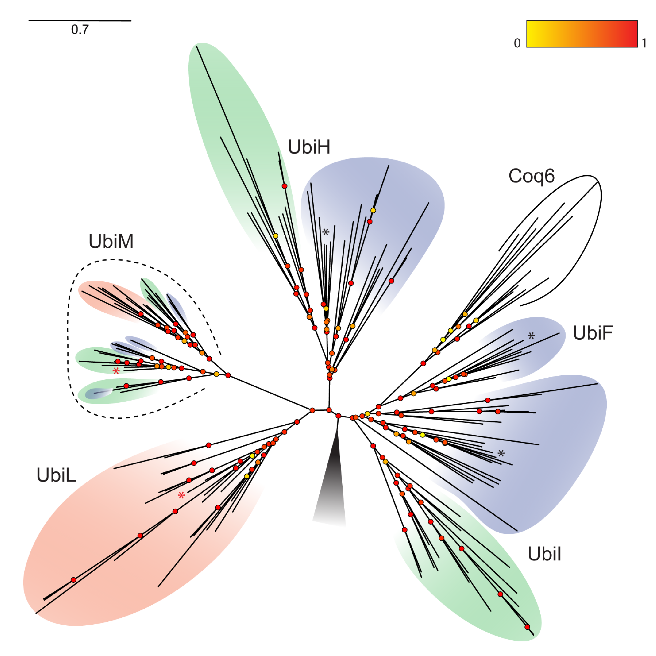
Figure 5 : Phylogénie des hydroxylases de la biosynthèse de Q chez les α, β et γ protéobactéries (respectivement, rose, vert et bleu).
Financements ↗
- 2022 Projet EUR-CBS « Ubicomplex »
- 2021 Projet Emergence-UGA (TIMC)
- 2020-2024 : ANR-19-CE44-0014 (projet O2-Taboo)
- 2020-2024 : ANR-19-CE45-0013 (projet Deepen; CNRS)
- 2015-2019 : ANR-15-CE11-0001 (projet (An)aeroUbi ; CNRS)
- 2014-2016: FRM (DPM20121125553)
Emplois et stages ↗
Venez travailler avec nous dans une atmosphère conviviale et productive. Nous sommes toujours prêts à travailler avec des étudiants et des post-docs motivés et intéressés par la biochimie, le métabolisme et l’évolution. Quel que soit le projet qui vous tente, contactez Fabien ou Ludovic par email (fabien.pierrel [at] univ-grenoble-alpes.fr ou ludovic.pelosi [at] univ-grenoble-alpes.fr).
Publications du groupe ↗
2022
Pierrel F., Burgardt A., Lee J.H., Pelosi L., Wendisch V.F. Recent progress on the roles, applications and microbial production of coenzyme Q. World Journal of Microbiology & Biotechnology 38 (2022) 58. DOI: 10.1007/s11274-022-03242-3
Saini P.K., Dawitz H., Aufschnaiter A., Bondarev S., Thomas J., Amblard A., Stewart J., Thierry-Mieg N., Ott M., Pierrel F. The [PSI+] prion modulates cytochrome c oxidase deficiency caused by deletion of COX12. Molecular Biology of the Cell (2022) 33. DOI : 10.1091/mbc.E21-10-0499
Diessl J., Berndtsson J., Broeskamp F., Habernig L., Kohler V., Vazquez-Calvo C., Nandy A., Peselj C., Drobysheva S., Pelosi L., Vögtle F-N., Pierrel F., Ott M., Büttner S. Manganese-driven CoQ deficiency. Nature Communications (2022) 13:6061. DOI : 10.1038/s41467-022-33641-x
Launay R., Teppa L., Martins C., Abby S., Pierrel F., André I., Esque J. Towards Molecular Understanding of the Functional Role of UbiJ-UbiK2 Complex in Ubiquinone Biosynthesis by Multiscale Molecular Modelling Studies. International Journal of Molecular Sciences (2022) 23:10323. DOI : 10.3390/ijms231810323
Schulz V, Basu S, Freibert SA, Webert H, Boß L, Mühlenhoff U, Pierrel F, Essen LO, Warui DM, Booker S, Stehling O, Lill R. Functional spectrum and specificity of mitochondrial ferredoxins FDX1 and FDX2. Nature Chemical Biology (2022) in press. DOI : 10.1038/s41589-022-01159-4
Burgardt A, Pelosi L, Hajj Chehade M, Wendisch VF*, Pierrel F*. Rational Engineering of Non-Ubiquinone Containing Corynebacterium glutamicum for Enhanced Coenzyme Q10 Production. Metabolites (2022) 12:428. DOI : 10.3390/metabo12050428
Arias-Cartín R, Uzel A, Seduk F, Gerbaud G, Pierrel F, Broc M, Lebrun R, Guigliarelli B, Magalon A, Grimaldi S, Walburger A. Identification and characterization of a noncanonical menaquinone-linked formate dehydrogenase. The Journal of Biological Chemistry (2022) 298:101384. DOI : 10.1016/j.jbc.2021.101384
2021
Kazemzadeh K., Hajj Chehade M., Hourdoir G., Brunet C.D., Caspar C., Loiseau L., Barras F., Pierrel P., Pelosi L. The biosynthetic pathway of ubiquinone contributes to pathogenicity of Francisella novicida. Journal of Bacteriology 203 (2021), e0040021. DOI: 10.1128/JB.00400-21
2020
Abby S.S. Kazemzadeh K., Vragnau C., Pelosi L.*, Pierrel F*. Advances in bacterial pathways for the biosynthesis of ubiquinone. Biochimica et Biophysica Acta (BBA)-Bioenergetics 1861 (2020) 148259. Co-corresponding auteur. DOI: 10.1016/j.bbabio.2020.148259
Vo* C.-D.T., Michaud* J., Elsen S., Faivre B., Bouveret E., Fontecave M., Pierrel F., Lombard M., Pelosi L. The O2-independent pathway of ubiquinone biosynthesis is essential for denitrification in Pseudomonas aeruginosa. The Journal of Biological Chemistry 292 (2020) 11937-11950. DOI: 10.1074/jbc.RA120.013748
2019
Pelosi* L., Vo* C.-D. T. Abby S. S. Loiseau L., Rascalou B., Hajj Chehade M., Faivre B., Goussé M., Chenal C., Touati T., Binet L., Cornu D., Fyfe C. D. Fontecave M., Barras F., Lombard M., Pierrel F. Ubiquinone biosynthesis over the entire O2 range: characterization of a conserved O2-independent pathway. mBIO 4, (2019) e01319-19. *Equal contribution. DOI: 10.1128/mBio.01319-19
Hajj Chehade M., Pelosi L., Fyfe C. D., Loiseau L., Rascalou B., Brugiere S., Kazemzadeh K., Vo C.-D. T., Ciccone L., Aussel L., Coute Y., Fontecave M., Barras, F., Lombard M., Pierrel, F. A Soluble metabolon ynthesizes the isoprenoid lipid ubiquinone. Cell Chemical Biology 26, (2019) 482-492. DOI: 10.1016/j.chembiol.2018.12.001
Acosta Lopez M. J., Trevisson E., Canton M., Vazquez-Fonseca L., Morbidoni V., Baschiera E., Frasson C., Pelosi L., Rascalou B., Desbats M. A., Alcázar-Fabra M., Ríos J. J., Sánchez-García A., Basso G., Navas P., Pierrel F., Brea-Calvo G., Salviati L. Vanillic acid restores coenzyme Q biosynthesis and ATP production in human cells lacking COQ6. Oxidative Medicine and Cellular Longevity 19, (2019) 3904905. DOI: 10.1155/2019/3904905
Szyttenholm, J., Chaspoul, F., Bauzan, M., Ducluzeau, A-L., Hajj Chehade, M., Pierrel, F., Denis, Y., Nitschke, W., Schoepp-Cothenet, B. The controversy on the ancestral arsenite oxidising enzyme; deducing evolutionary histories with phylogeny and thermodynamics. BBA Bioenergetics (2020) 1861:148252. DOI : 10.1016/j.bbabio.2020.148252
Fino, C., Vestergaard, M., Ingmer, H., Pierrel, F., Gerdes, K., Harms., A. PasT of Escherichia coli sustains antibiotic tolerance and aerobic respiration as a bacterial homolog of mitochondrial Coq10. Microbiology Open (2020) 9:e1064. DOI : 10.1002/mbo3.1064
Cerqua, C., Casarin, A., Pierrel, F., Vazquez Fonseca, L., Viola, G., Salviati, L., Trevisson, E. Vitamin K2 cannot substitute Coenzyme Q10 as electron carrier in the mitochondrial respiratory chain of mammalian cells. Scientific Reports (2019) 9:6553. DOI : 10.1038/s41598-019-43014-y
2017
Loiseau L, Fyfe C, Aussel L, Hajj Chehade M, Hernández SB, Faivre B, Hamdane D, Mellot-Draznieks C., Rascalou B., Pelosi L., Velours C., Cornu D., Lombard M., Casadesús J., Pierrel F., Fontecave M., Barras F. The UbiK protein is an accessory factor necessary for bacterial ubiquinone (UQ) biosynthesis and forms a complex with the UQ biogenesis factor UbiJ. The Journal of Biological Chemistry 292, (2017) 11937-11950. DOI: 10.1074/jbc.M117.789164
Pierrel, F. Impact of chemical analogs of 4-Hydroxybenzoic acid on coenzyme Q biosynthesis: from inhibition to bypass of coenzyme Q deficiency. Frontiers in Physiology 8, (2017). DOI: 10.3389/fphys.2017.00436
2016
Pelosi* L., Ducluzeau* A.L., Loiseau L., Barras F., Schneider D., Junier I., Pierrel F. Evolution of ubiquinone biosynthesis: multiple proteobacterial enzymes with various regioselectivities to catalyze three contiguous aromatic hydroxylation reactions. mSystems. 1, (2016) (4). e00091-16. *Equal contribution. DOI: 10.1128/mSystems.00091-16
Payet* L.A., Leroux* M., Willison J.C., Kihara A, Pelosi L., Pierrel F. Mechanistic details of early steps in coenzyme Q biosynthesis pathway in yeast., Cell Chemical Biology 23, (2016) 1241-1250. * Equal contribution. DOI: 10.1016/j.chembiol.2016.08.008
Stefely J. A., Licitra F., Laredj L., Reidenbach A. G., Kemmerer Z. A., Grangeray A., … , Pierrel F., Zoll J., Dal Peraro M., Kannan N., Bingman C. A., Coon J. J., Isope P., Puccio H., Pagliarini D. J. Cerebellar Ataxia and Coenzyme Q Deficiency through Loss of Unorthodox Kinase Activity. Molecular Cell (2016) 63: 608-20. DOI : 10.1016/j.molcel.2016.06.030
Desbats M. A., Morbidoni V., Silic-Benussi M., Doimo M., Ciminale V., Cassina M., Sacconi S., Hirano M., Basso G., Pierrel F., Navas P., Salviati L., Trevisson E. The COQ2 genotype predicts the severity of coenzyme Q10 deficiency. Hum Mol Genet. (2016) 25:4256-4265. DOI: 10.1093/hmg/ddw257
Ismail A., Leroux V., Smadja M., Gonzalez L., Lombard M., Pierrel F., Mellot-Draznieks C., Fontecave M. Coenzyme Q Biosynthesis: Evidence for a Substrate Access Channel in the FAD-Dependent Monooxygenase Coq6. PLoS Comput Biol (2016) 12: e1004690. DOI: 10.1371/journal.pcbi.1004690
2015
Ozeir M., Pelosi L., Ismail A., Mellot-Draznieks C., Fontecave M., Pierrel F. Coq6 is responsible for the C4-deamination reaction in coenzyme Q biosynthesis in Saccharomyces cerevisiae, The Journal of Biological Chemistry 290, (2015) 24140-24151. DOI: 10.1074/jbc.M115.675744
Rendon J., Pilet E., Fahs Z., Seduk F., Sylvi L., Hajj Chehade M., Pierrel F., Guigliarelli B., Magalon A., Grimaldi S. Demethylmenaquinol is a substrate of Escherichia coli nitrate reductase A (NarGHI) and forms a stable semiquinone intermediate at the NarGHI quinol oxidation site. Biochim Biophys Acta (2015) 1847:739-747. DOI:10.1016/j.bbabio.2015.05.001
2014
Doimo, M., Trevisson, E., Airik, R., Bergdoll, M., Santos-Ocana, C., Hildebrandt, F., Navas, P., Pierrel, F., and Salviati, L. Effect of vanillic acid on COQ6 mutants identified in patients with coenzyme Q deficiency. Biochim. Biophys. Acta 1842, (2014) 1-6. DOI: 10.1016/j.bbadis.2013.10.007
Aussel, L., Loiseau, L., Hajj Chehade, M., Pocachard, B., Fontecave, M., Pierrel, F., and Barras, F. ubiJ, a New gene required for aerobic growth and proliferation in macrophage, is involved in coenzyme Q biosynthesis in Escherichia coli and Salmonella enterica serovar Typhimurium. J. Bacteriol. 196, (2014) 70-79 DOI: 10.1128/JB.01065-13
2013
Hajj Chehade, M., Loiseau, L., Lombard, M., Pecqueur, L., Ismail, A., Smadja, M., Golinelli-Pimpaneau, B., Mellot-Draznieks, C., Hamelin, O., Aussel, L., Kieffer-Jaquinod, S., Labessan, N., Barras, F., Fontecave, M., and Pierrel, F. ubiI, a Nnew gene in Escherichia coli coenzyme Q biosynthesis, is involved in aerobic C5-hydroxylation. J. Biol. Chem. 288, (2013) 20085-20092. DOI: 10.1074/jbc.M113.480368
2012
Xie, L. X., Ozeir, M., Tang, J. Y., Chen, J. Y., Kieffer-Jaquinod, S., Fontecave, M., Clarke, C. F., and Pierrel, F. Over-expression of the Coq8 kinase in Saccharomyces cerevisiae coq null mutants allows for accumulation of diagnostic intermediates of the Coenzyme Q6 biosynthetic pathway. J. Biol. Chem. 287, (2012) 23571-23581 DOI: 10.1016/j.bbalip.2015.05.003
2011
Ozeir, M., Muhlenhoff, U., Webert, H., Lill, R., Fontecave, M., and Pierrel, F. Coenzyme Q biosynthesis: Coq6 Is required for the C5-hydroxylation reaction and substrate analogs rescue Coq6 deficiency. Chem. Biol. 18, (2011) 1134-1142 DOI: 10.1016/j.chembiol.2011.07.008
2010
Pierrel, F., Hamelin, O., Douki, T., Kieffer-Jaquinod, S., Muhlenhoff, U., Ozeir, M., Lill, R., and Fontecave, M. Involvement of mitochondrial ferredoxin and para-aminobenzoic acid in yeast coenzyme Q biosynthesis. Chem. Biol. 17, (2010) 449-459 DOI: 10.1016/j.chembiol.2010.03.014
